[Porto Seguro] XGBoost_CV(LB.284)
[공지사항] “출처: https://www.kaggle.com/code/jundthird/kor-xgboost-cv-lb-284”
MAX_ROUNDS = 400
OPTIMIZE_ROUNDS = False
LEARNING_RATE = 0.07
EARLY_STOPPING_ROUNDS = 10
# EARLY_STOPPING_ROUNDS를 매우 높게 설정하였습니다.(50, OPTIMIZE_ROUNDS가 설정되어 있을 때)
# 실제로 early stopping을 사용하고 싶다면 EARLY_STOPPING_ROUNDS를 줄여주세요
rounds의 적절한 수에 대한 아이디어를 얻기 위해 초기에 MAX_ROUNDS를 매우 높게 설정하고 OPTIMIZE_ROUNDS를 사용하는 것을 추천합니다. (모든 fold 중에서 $best\_ntree\_limit$의 최댓값에 가까워야하며 모델이 적절하게 정규화되었다면 조금 더 높을 수 있습니다. 아니면 $verbose=True$로 설정하여 디테일을 살펴본 후 모든 fold에 잘 작동되는 round를 찾습니다). 그 후, OPTIMIZEZ_ROUNDS를 끄고, 최적의 MAX_ROUNDS의 값을 설정합니다.
각 fold에서 가장 적합한 round를 설정해 early stopping하는 가장 큰 문제점은 validation data에 overfitting된다는 것입니다. 그러므로 test data를 예측하는데 최적의 모델을 만들지 못할 가능성이 있고, 만약 다른 모델과의 stacking/ensembling을 위한 validation data를 생성하는데 사용된다면 이 모델이 앙상블에 너무 많은 weight을 갖게 됩니다. 또 다른 가능성(XGBoost의 default)은 최적의 round보다 early stopping이 일어났을때의 round를 사용한다는 것입니다.
early stopping이 overfitting 문제는 해결하지만, 20-round early stopping에서 일정한 값의 round보다 validation score가 낮게 나온 것을 봤을때, early stopping은 약간 underfitting된 것처럼 보입니다.
import numpy as np
import pandas as pd
from xgboost import XGBClassifier
from sklearn.model_selection import train_test_split
from sklearn.model_selection import StratifiedKFold
from sklearn.preprocessing import LabelEncoder
from numba import jit
import time
import gc
# Compute gini
@jit # 넘파이 코드를 빠르게 실행시켜주는 JIT 컴파일러
def eval_gini(y_true, y_prob):
y_true = np.asarray(y_true) # numba가 이해할 수 있는 형식으로 변환
y_true = y_true[np.argsort(y_prob)]
ntrue = 0
gini = 0
delta = 0
n = len(y_true)
for i in range(n-1, -1, -1):
y_i = y_true[i]
ntrue += y_i
gini += y_i * delta
delta += 1 - y_i
gini = 1 - 2 * gini / (ntrue * (n - ntrue))
return gini
def gini_xgb(preds, dtrain):
labels = dtrain.get_label()
gini_score = -eval_gini(labels, preds)
# 해당 커널에서 분류에 사용한 XGBClassifier모델은 평가 지표로 rmse와 같은 값을 사용 -> 즉 오류의 최솟값을 찾음
# 이 대회의 평가 지표인 지니 계수 : 0.5에 가까울수록(값이 클수록) 좋은 값이기 때문에 -를 붙여주는 함수를 생성
return [("gini", gini_score)]
범주형 변수 인코딩
해당 커널에서는 distinct value가 많은 ps_car_11_cat 변수들에 대해서는 mean encoding을 사용하였고, 나머지 범주형 변수들에 대해서는 더미 변수를 생성하는 one-hot encoding의 방식 사용
Mean encoding이란?
- 목표
카테고리 변수에 대하여 (여기서는 104개의 카테고리를 가진 ps_car_11_cat 변수에 대하여) 단순하게 0,1로 구분된 target값에 대한 의미를 가지도록 만드는 것
- Method
카테고리 변수의 Label 값에 따라서 Target 값의 평균을 구해 각 Label이 Target과 가지는 상관성, 영향 도출
-
문제점
-
target값을 이용해 계산하기 때문에 overfitting의 문제가 발생할 수 있음 -> 이 커널에서는 noise를 추가하는 방식으로 이 문제를 해결
-
test 데이터와 train 데이터 간의 분포가 다른 경우 (ex. 한쪽이 불균형 데이터인 경우) 이때도 마찬가지로 overfitting의 문제 발생 가능 -> Smoothing을 통해 문제 해결
-
Smoothing 공식
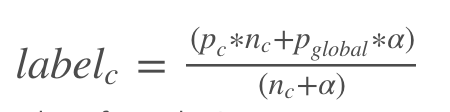
def add_noise(series, noise_level):
return series * (1 + noise_level * np.random.randn(len(series)))
def target_encode(trn_series=None,
val_series=None,
tst_series=None,
target=None,
min_samples_leaf=1,
smoothing=1,
noise_level=0):
'''
trn_series : training categorical feature as a pd.Series
tst_series : test categorical feature as a pd.Series
target : target data as a pd.Series
min_samples_leaf (int): category 평균을 고려하기 위한 최소 샘플 수
smoothing (int): categorical average와 prior의 균형을 맞추기 위한 smoothing effect
'''
assert len(trn_series) == len(target)
assert trn_series.name == tst_series.name
temp = pd.concat([trn_series, target], axis=1)
# Compute target mean
averages = temp.groupby(by=trn_series.name)[target.name].agg(["mean", "count"])
# Compute smoothing
smoothing = 1 / (1 + np.exp(-(averages["count"] - min_samples_leaf) / smoothing))
# Apply average function to all target data
prior = target.mean()
# The bigger the count the less full_avg is taken into account
averages[target.name] = prior * (1 - smoothing) + averages["mean"] * smoothing
averages.drop(["mean", "count"], axis=1, inplace=True)
# Apply averages to trn and tst series
ft_trn_series = pd.merge(
trn_series.to_frame(trn_series.name),
averages.reset_index().rename(columns={'index': target.name, target.name: 'average'}),
on=trn_series.name,
how='left')['average'].rename(trn_series.name + '_mean').fillna(prior)
# pd.merge does not keep the index so restore it
ft_trn_series.index = trn_series.index
ft_val_series = pd.merge(
val_series.to_frame(val_series.name),
averages.reset_index().rename(columns={'index': target.name, target.name: 'average'}),
on=val_series.name,
how='left')['average'].rename(trn_series.name + '_mean').fillna(prior)
# pd.merge does not keep the index so restore it
ft_val_series.index = val_series.index
ft_tst_series = pd.merge(
tst_series.to_frame(tst_series.name),
averages.reset_index().rename(columns={'index': target.name, target.name: 'average'}),
on=tst_series.name,
how='left')['average'].rename(trn_series.name + '_mean').fillna(prior)
# pd.merge does not keep the index so restore it
ft_tst_series.index = tst_series.index
return add_noise(ft_trn_series, noise_level), add_noise(ft_val_series, noise_level), add_noise(ft_tst_series, noise_level)
train_df = pd.read_csv('./porto_seguro/train.csv', na_values="-1")
test_df = pd.read_csv('./porto_seguro/test.csv', na_values="-1")
# from olivier
train_features = [
"ps_car_13", # : 1571.65 / shadow 609.23
"ps_reg_03", # : 1408.42 / shadow 511.15
"ps_ind_05_cat", # : 1387.87 / shadow 84.72
"ps_ind_03", # : 1219.47 / shadow 230.55
"ps_ind_15", # : 922.18 / shadow 242.00
"ps_reg_02", # : 920.65 / shadow 267.50
"ps_car_14", # : 798.48 / shadow 549.58
"ps_car_12", # : 731.93 / shadow 293.62
"ps_car_01_cat", # : 698.07 / shadow 178.72
"ps_car_07_cat", # : 694.53 / shadow 36.35
"ps_ind_17_bin", # : 620.77 / shadow 23.15
"ps_car_03_cat", # : 611.73 / shadow 50.67
"ps_reg_01", # : 598.60 / shadow 178.57
"ps_car_15", # : 593.35 / shadow 226.43
"ps_ind_01", # : 547.32 / shadow 154.58
"ps_ind_16_bin", # : 475.37 / shadow 34.17
"ps_ind_07_bin", # : 435.28 / shadow 28.92
"ps_car_06_cat", # : 398.02 / shadow 212.43
"ps_car_04_cat", # : 376.87 / shadow 76.98
"ps_ind_06_bin", # : 370.97 / shadow 36.13
"ps_car_09_cat", # : 214.12 / shadow 81.38
"ps_car_02_cat", # : 203.03 / shadow 26.67
"ps_ind_02_cat", # : 189.47 / shadow 65.68
"ps_car_11", # : 173.28 / shadow 76.45
"ps_car_05_cat", # : 172.75 / shadow 62.92
"ps_calc_09", # : 169.13 / shadow 129.72
"ps_calc_05", # : 148.83 / shadow 120.68
"ps_ind_08_bin", # : 140.73 / shadow 27.63
"ps_car_08_cat", # : 120.87 / shadow 28.82
"ps_ind_09_bin", # : 113.92 / shadow 27.05
"ps_ind_04_cat", # : 107.27 / shadow 37.43
"ps_ind_18_bin", # : 77.42 / shadow 25.97
"ps_ind_12_bin", # : 39.67 / shadow 15.52
"ps_ind_14", # : 37.37 / shadow 16.65
]
# add combinations
combs = [
('ps_reg_01', 'ps_car_02_cat'),
('ps_reg_01', 'ps_car_04_cat'),
]
# Process data
id_test = test_df['id'].values
id_train = train_df['id'].values
y = train_df['target']
start = time.time()
for n_c, (f1, f2) in enumerate(combs):
name1 = f1 + "_plus_" + f2
print('current feature %60s %4d in %5.1f'
% (name1, n_c + 1, (time.time() - start) / 60))
train_df[name1] = train_df[f1].apply(lambda x: str(x)) + "_" + train_df[f2].apply(lambda x: str(x))
test_df[name1] = test_df[f1].apply(lambda x: str(x)) + "_" + test_df[f2].apply(lambda x: str(x))
# Label Encode
lbl = LabelEncoder()
lbl.fit(list(train_df[name1].values) + list(test_df[name1].values))
train_df[name1] = lbl.transform(list(train_df[name1].values))
test_df[name1] = lbl.transform(list(test_df[name1].values))
train_features.append(name1)
X = train_df[train_features]
test_df = test_df[train_features]
f_cats = [f for f in X.columns if "_cat" in f]
current feature ps_reg_01_plus_ps_car_02_cat 1 in 0.0
current feature ps_reg_01_plus_ps_car_04_cat 2 in 0.0
y_valid_pred = 0 * y
y_test_pred = 0
# Set up folds
K = 5
kf = StratifiedKFold(n_splits=K, random_state=1, shuffle=True)
np.random.seed(0)
# Set up classifier
model = XGBClassifier(
n_estimators=MAX_ROUNDS,
max_depth=4,
objective="binary:logistic",
learning_rate=LEARNING_RATE,
subsample=.8,
min_child_weight=6,
colsample_bytree=.8,
scale_pos_weight=1.6,
gamma=10,
reg_alpha=8,
reg_lambda=1.3,
)
# Run CV
for i, (train_index, test_index) in enumerate(kf.split(train_df, train_df["target"])):
# Create data for this fold
y_train, y_valid = y.iloc[train_index].copy(), y.iloc[test_index]
X_train, X_valid = X.iloc[train_index,:].copy(), X.iloc[test_index,:].copy()
X_test = test_df.copy()
print( "\nFold ", i)
# Enocode data
for f in f_cats:
X_train[f + "_avg"], X_valid[f + "_avg"], X_test[f + "_avg"] = target_encode(
trn_series=X_train[f],
val_series=X_valid[f],
tst_series=X_test[f],
target=y_train,
min_samples_leaf=200,
smoothing=10,
noise_level=0
)
# Run model for this fold
if OPTIMIZE_ROUNDS:
eval_set=[(X_valid,y_valid)]
fit_model = model.fit( X_train, y_train,
eval_set=eval_set,
eval_metric=gini_xgb,
early_stopping_rounds=EARLY_STOPPING_ROUNDS,
verbose=False
)
print( " Best N trees = ", model.best_ntree_limit )
print( " Best gini = ", model.best_score )
else:
fit_model = model.fit( X_train, y_train )
# Generate validation predictions for this fold
pred = fit_model.predict_proba(X_valid)[:,1]
# predict_proba의 출력은 각 클래스에 대한 확률, 이진 분류에서는 항상 사이즈가 (n_samples, 2)
print(" Gini = ", eval_gini(y_valid, pred))
y_valid_pred.iloc[test_index] = pred
# Accumulate test set predictions
y_test_pred += fit_model.predict_proba(X_test)[:, 1]
del X_test, X_train, X_valid, y_train
y_test_pred /= K
print( "\nGini for full training set:" )
eval_gini(y, y_valid_pred)
Fold 0
Gini = 0.2782370292877412
Fold 1
Gini = 0.280692079296757
Fold 2
Gini = 0.2867285610185394
Fold 3
Gini = 0.2864232547656165
Fold 4
Gini = 0.2918269435375388
Gini for full training set:
0.2847372930699126
# Save validation predictions for stacking/ensembling
val = pd.DataFrame()
val['id'] = id_train
val['target'] = y_valid_pred.values
val.to_csv('xgb_valid.csv', float_format='%.6f', index=False)
# Create submission file
sub = pd.DataFrame()
sub['id'] = id_test
sub['target'] = y_test_pred
sub.to_csv('xgb_submit.csv', float_format='%.6f', index=False)
참고
댓글남기기